Tumorheterogenität bei schwer behandelbaren Krebsarten im Fokus
Im Rahmen der Dekade gegen Krebs werden Pionierarbeiten gefördert, die die Tumorheterogenität und die sich daraus ergebende Therapieresistenz von 3 häufigen Krebsarten in den Fokus nehmen. Ziel ist die Weiterentwicklung von Therapieansätzen.

Das zur Förderung empfohlene Konsortium „SATURN3“ wird die räumliche und zeitliche Entwicklung der „Tumorheterogenität“ in Krebserkrankungen im Detail untersuchen. „Tumorheterogenität“ bezeichnet die Verschiedenartigkeit von Zellen innerhalb eines Tumors, die bei Resistenz- und Metastasenbildung sowie bei einem Rückfall eine bedeutende Rolle spielt. Die beteiligten Forscherinnen und Forscher konzentrieren sich dabei auf drei davon besonders betroffene Formen des Brust-, Darm- und Bauchspeicheldrüsenkrebs (s. Kasten).
Das langfristige Ziel von SATURN3 ist die Weiterentwicklung erfolgversprechender Therapieansätze. Diese zielen auf die Prozesse der Tumorheterogenität, um die Überlebenschancen von Menschen zu erhöhen, die an diesen bislang schwer behandelbaren Tumoren leiden.
Was hat Tumorheterogenität mit Therapieresistenz zu tun?
Die auf verschiedenen Ebenen (u.a. genetisch, epigenetisch und molekular) veränderten Krebszellen befähigen den Tumor auf diverse Arten zum Wachsen.
Dabei entwickeln Tumorzellen unterschiedliche Eigenschaften, die einzelne Zellen oder Zellgruppen (sog. Zellklone) zur Metastasierung sowie zur Resistenz gegenüber Therapien befähigen. Die Zellen erlangen dadurch neue biologische Eigenschaften, was man als Tumor-Evolution bezeichnet. Das macht es schwer, alle Zellen eines Tumors mit nur einer Therapie angreifen zu können.
Um dem besser entgegenzuwirken, will das SATURN3-Konsortium nun das Zusammenspiel der entscheidenden Faktoren im Detail aufklären.
Die Forschenden nutzen bei ihren Arbeiten innovative Analysemethoden wie beispielsweise flüssigkeits- und bildbasierte Überwachung auf Einzelzell-Ebene.
Nutzung modernster Analysemethoden

Eine wichtige Säule bilden dabei die „Omics-Technologien“, die es erlauben die Veränderungen in Krebszellen zu ermitteln. Frühere Omics-Ansätze waren nur geeignet, viele Zellen auf einmal zu untersuchen und daraus gemittelte Ergebnisse anzuzeigen. So können die Analyseergebnisse jedoch nicht einzelnen Zellen oder Bereichen zugeordnet und damit die Heterogenität von Tumoren nicht ausreichend dargestellt werden. Durch die neue Methode der Einzelzell-Sequenzierung lassen sich inzwischen gezielt auch einzelne Krebszellen aus den verschiedenen Tumorbereichen (den „Tumorzellklonen“) analysieren. Damit erhalten die Wissenschaftlerinnen und Wissenschaftler nun erstmals ein zeitaufgelöstes, mehrdimensionales Bild von Tumoren auf Einzelzell-Ebene.
Minimal-invasiv für die Betroffenen
Den Patientinnen und Patienten werden dafür auf schonende Art Zellen aus verschiedenen Tumorbereichen (z. B. aus verschiedenen Metastasen und dem Primärtumor) zu Beginn der personalisierten Diagnostik entnommen und die Veränderungen auf den Ebenen bestimmt, von denen man weiß, dass sie einen Einfluss auf das Krebsgeschehen haben.
Zudem werden für das Projekt wiederholt Proben aus einzelnen Tumorbereichen zu verschiedenen Zeitpunkten der Erkrankung entnommen und vergleichend analysiert (longitudinale Probengewinnung), um das Fortschreiten der Veränderungen zu sehen. So wollen die Wissenschaftlerinnen und Wissenschaftler erstmals die Mechanismen der Tumorheterogenität während des gesamten Krankheitsverlaufs in verschiedenen Zellklonen verfolgen.
Was sich durch die Omics-Analysen herausfinden lässt und was nicht
Zeitlichen Ablauf bei der Entwicklung von Tumorheterogenität ermitteln
Die Konsortialpartner nutzen bereits laufende Studien (unter anderem die am Nationalen Centrum für Tumorerkrankungen (NCT) durchgeführten Studien CATCH und COGNITION), in denen zu verschiedenen Zeiten bereits Gewebeproben und Flüssigbiopsien einer Erkrankung entnommen und eingelagert wurden.
Anhand dieser Proben vergleichen die Forschenden Zellen und Zellbestandteile (insbesondere DNA und RNA) aus verschiedenen Bereichen des Tumors, die den Betroffenen jeweils vor und nach der Therapie, vor und nach einem Rückfall und vor und nach einer Metastasierung entnommen wurden. Damit lassen sich die Veränderungen, die in den verschiedenen Zellklonen im Verlauf der Erkrankung aufgetreten sind, zeitlich nachverfolgen und z.B. dem Einfluss der Therapie zuordnen.
Mehr zu CATCH und COGNITION (Nationales Centrum für Tumorerkrankungen Heidelberg)
Big Data auswerten
Omics-Untersuchungen erzeugen sehr komplexe und große Datenmengen (man spricht von „Big Data“). Um diese optimal zu analysieren, werden die Forschenden modernste Analysemethoden und computergestützte Auswerteverfahren entwickeln. Dafür bedienen sie sich bioinformatischer Methoden – unter anderem auch unter Einsatz von Künstlicher Intelligenz (KI).
Die Prozesse der Tumorheterogenitätsentwicklung im Detail aufklären
Mit Omics-Analysen lassen sich vor allem das Vorkommen und die Mengenverhältnisse von z.B. Aufklärung der vielfältigen Veränderungen in Krebszellen zu einem bestimmten Zeitpunkt feststellen.
Die Forschenden wollen auch nachvollziehen, welche Signale das Fortschreiten von Tumorheterogenität anzeigen, wie die Signale innerhalb der Zellen vermittelt werden und welche Faktoren (z.B. Einflüsse aus der Tumor-Mikroumgebung) das Geschehen im weiteren Verlauf beeinflussen. Um die Zwischenschritte des Krebsentstehungsprozesses und den räumlichen Verlauf (also an welchen Stellen des Tumors diese zuerst stattfinden) der Veränderungen mitzuverfolgen, werden in dem Projekt ergänzende Methoden zum Einsatz kommen.

Molekulare Bildgebung
Unter anderem wird die innovative Molekulare Bildgebung genutzt. Diese macht Stoffwechselprozesse und Eigenschaften des Tumormikromilieus innerhalb des Körpers sichtbar und stellt sie im räumlichen Verlauf dar.
Wie funktioniert Molekulare Bildgebung
An (Signal-)Proteine, die zu den individuellen Wachstumseigenschaften von Tumorzellen gehören, werden Marker gekoppelt, die sie in der Bildgebung sichtbar machen. So lässt sich sehen, in welchen Bereichen des Tumors bzw. den Metastasen sich das entsprechende Protein anhäuft. Daraus kann man Rückschlüsse über die in den unterschiedlichen Tumorzellklonen ablaufenden Signalwege und damit verbundene biologische Eigenschaften dieser Zellen/Zellklone ziehen.
Auch diese Methode hat ihre Begrenzung: Patientinnen bzw. Patienten können nicht dauerhaft mittels Bildgebung durchleuchtet werden, um den Krankheitsverlauf genau zu erfassen. Stellvertretend für die Erkrankungen werden daher ebenso innovative „Organoid“-Modelle genutzt.
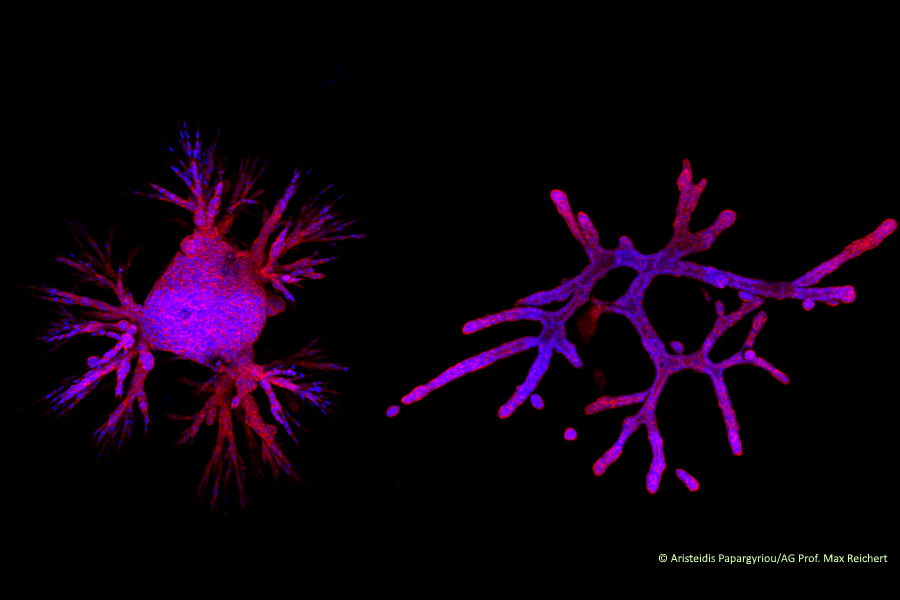
Wie funktionieren Organoid-Modelle
Das sind Minitumoren, die in der Petrischale aus den Krebszellen von Betroffenen herangezüchtet werden. So lassen sich aus vielen unterschiedlichen Tumorarealen generierte „Avatare“ der Tumoren gewinnen, an denen die biologischen Prozesse außerhalb des Betroffenen untersucht und zudem weitere Tests durchgeführt werden können. Das ermöglicht noch viel umfassendere Möglichkeiten, die Ursachen und Zusammenhänge zwischen molekularen Mechanismen zu bestimmen, die für die Fortentwicklung der Erkrankung verantwortlich sind.
An den Organoiden lassen sich Einflüsse und Reaktionen der Zellen auf Behandlungsformen simulieren, beispielsweise Medikamente testen und einkreisen, welche Therapieform bei den vorliegenden Veränderungen am effektivsten sind – aber auch, inwieweit bestimmte eingesetzte Therapien womöglich zum Antreiber einer Resistenzbildung werden.
Wie die Daten den Betroffenen zugute kommen
Mit den Daten vieler Erkrankter lässt sich eine Kartierung vornehmen, wie sich die Krebszellen zwischen verschiedenen Regionen im Primärtumor und den Metastasen über den zeitlichen Verlauf der Erkrankung hinweg auseinanderentwickeln und welche Faktoren darauf einen Einfluss haben. Auf der Basis dieser Daten lassen sich Wege finden, um die für den Patienten ungünstige Heterogenitätsentwicklung therapeutisch zu adressieren. Aus den gewonnenen Erkenntnissen werden zudem Vorhersagemodelle entwickelt, sodass eine Therapieresistenz bei Patientinnen und Patienten frühzeitig aufgedeckt werden kann.
Anhand dieser Erkenntnisse werden die Forscherinnen und Forscher verlässliche Biomarker identifizieren die es ermöglichen die besten therapeutischen Strategien für den klinischen Einsatz anhand einer Vorselektion von Patienten wählen zu können. Diese Biomarker können zum Beispiel in einer Blutprobe des Betroffenen detektiert werden und u.a. Therapiebegleitend den Behandlungserfolg und eine einsetzende Resistenzbildung monitoren.
Das Konsortium umfasst Forschende
der Universitäten:
- Essen
- Heidelberg
- Frankfurt
- Freiburg
- Köln
- Marburg
- Augsburg
- Regensburg
- München (TU und LMU)
- Göttingen
sowie des
- Deutschen Krebsforschungszentrums (DKFZ)
- Europäischen Laboratoriums für Molekularbiologie - Heidelberg (EMBL)
- des Georg Speyer Hauses
- der HI-STEM gGmbH
- der GBG ForschungsGmbH
Koordinator ist Prof. Dr. Jens Siveke. Er leitet die Abteilung für Translationale Onkologie Solider Tumore (ein Standort des Deutschen Konsortium für Translationale Krebsforschung, DKTK) am Westdeutschen Tumorzentrum Essen (WTZ) und ist Direktor des Brückeninstituts für Experimentelle Tumortherapie (BIT).
Die Erkenntnisse aus diesen umfassenden Forschungsarbeiten werden zusammen mit pharmazeutischen Partnern in Studienprotokolle für patientenzentrierte Therapien überführt, um die erarbeiteten Prinzipien gegen fortgeschrittenen oder schwer behandelbaren Krebs schnellstmöglich in die klinische Anwendung zu bringen.